血小板减少异质性分析
问题背景
探究脓毒症相关的血小板减少异质性
解决方案
利用MIMIC-IV数据库、e-ICU数据库等其他数据库数据,使用机器学习方法探究脓毒症相关血小板减少亚型 首先进行数据提取
数据提取
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
332
333
334
335
336
337
338
339
340
341
342
343
344
345
346
347
348
349
350
351
352
353
354
355
356
357
358
359
360
361
362
363
364
365
366
367
368
369
370
371
372
373
374
375
376
377
378
379
380
381
382
383
384
385
386
387
388
389
390
391
392
393
394
395
396
397
398
399
400
401
402
403
404
405
406
407
408
409
410
411
412
413
414
415
416
417
418
419
420
421
422
423
424
425
426
427
428
429
430
431
432
433
434
435
436
437
438
439
440
441
442
443
444
445
446
447
448
449
450
451
452
453
454
455
456
457
458
459
460
461
462
463
464
465
466
467
468
469
470
471
472
473
474
475
476
477
478
479
480
481
482
# 导入必要的库
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sqlalchemy import create_engine
import psycopg2
import os
from datetime import datetime, timedelta
import warnings
warnings.filterwarnings('ignore')
# 设置绘图风格
plt.style.use('ggplot')
sns.set(style="whitegrid")
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文
plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号
# 数据库连接
def connect_to_postgres():
"""创建数据库连接"""
# 替换以下信息为您的PostgreSQL连接信息
conn_string = "postgresql://postgres:123456@localhost:1116/mimiciv"
engine = create_engine(conn_string)
conn = engine.connect()
return engine, conn
# 提取脓毒症患者队列
def extract_sepsis_cohort(engine):
"""
从派生表中提取Sepsis-3定义的脓毒症患者
参数:
engine: SQLAlchemy数据库引擎
返回:
sepsis_df: 包含脓毒症患者信息的DataFrame
"""
query = """
SELECT s.*,
p.gender, p.anchor_age as age,
a.hospital_expire_flag,
a.admittime, a.dischtime,
i.intime as icu_intime, i.outtime as icu_outtime,
i.los as icu_los
FROM mimiciv_derived.sepsis3 s
INNER JOIN mimiciv_icu.icustays i ON s.stay_id = i.stay_id
INNER JOIN mimiciv_hosp.admissions a ON i.hadm_id = a.hadm_id
INNER JOIN mimiciv_hosp.patients p ON s.subject_id = p.subject_id
"""
# 执行SQL查询并将结果存储到sepsis_df
sepsis_df = pd.read_sql(query, engine)
print(f"提取到 {len(sepsis_df)} 名脓毒症患者")
if 'hadm_id' not in sepsis_df.columns:
# 从icustays表获取stay_id到hadm_id的映射
query = """
SELECT DISTINCT stay_id, hadm_id
FROM mimiciv_icu.icustays
"""
stay_hadm_map = pd.read_sql(query, engine)
sepsis_df = sepsis_df.merge(stay_hadm_map, on='stay_id', how='left')
return sepsis_df
# 提取血小板计数数据
def extract_platelet_data(engine, sepsis_df):
"""
提取脓毒症患者的血小板计数数据
参数:
engine: SQLAlchemy数据库引擎
sepsis_df: 脓毒症患者DataFrame
返回:
plt_df: 血小板计数数据
"""
# 获取所有脓毒症患者的stay_id列表
stay_ids = tuple(sepsis_df['stay_id'].unique())
# 如果只有一个ID,需要特殊处理tuple语法
if len(stay_ids) == 1:
stay_ids_str = f"({stay_ids[0]})"
else:
stay_ids_str = str(stay_ids)
# 查询血小板计数数据
# MIMIC-IV中血小板计数的ITEMID为51265
query = f"""
SELECT le.subject_id, le.hadm_id, i.stay_id,
le.charttime, le.valuenum as platelet_count
FROM mimiciv_hosp.labevents le
INNER JOIN mimiciv_icu.icustays i ON le.subject_id = i.subject_id
AND le.charttime BETWEEN i.intime - INTERVAL '24 HOURS' AND i.outtime + INTERVAL '24 HOURS'
WHERE i.stay_id IN {stay_ids_str}
AND le.itemid = 51265 -- 血小板计数的ITEMID
AND le.valuenum IS NOT NULL
AND le.valuenum > 0
AND le.valuenum < 1000 -- 排除异常值
ORDER BY le.subject_id, le.hadm_id, le.charttime
"""
plt_df = pd.read_sql(query, engine)
print(f"提取到 {len(plt_df)} 条血小板计数记录,涉及 {plt_df['stay_id'].nunique()} 位患者")
return plt_df
# 提取其他实验室检查数据
def extract_lab_data(engine, sepsis_df):
"""
提取脓毒症患者的其他关键实验室检查数据
参数:
engine: SQLAlchemy数据库引擎
sepsis_df: 脓毒症患者DataFrame
返回:
lab_df: 实验室检查数据
"""
# 获取所有脓毒症患者的stay_id列表
stay_ids = tuple(sepsis_df['stay_id'].unique())
# 如果只有一个ID,需要特殊处理tuple语法
if len(stay_ids) == 1:
stay_ids_str = f"({stay_ids[0]})"
else:
stay_ids_str = str(stay_ids)
# 关键实验室检查项目的ITEMID
lab_items = {
'50912': 'creatinine',
'50971': 'bilirubin_total',
'50983': 'sodium',
'50902': 'chloride',
'51300': 'wbc',
'51301': 'wbc_diff',
'51221': 'hematocrit',
'50931': 'glucose',
'50960': 'magnesium',
'50970': 'phosphate',
'51275': 'ptt',
'51237': 'inr',
'51274': 'pt',
'50861': 'alt',
'50878': 'ast',
'50885': 'bun',
'50976': 'lactate',
'51491': 'troponin',
'50889': 'calcium'
}
# 构建ITEMID筛选条件
item_ids_str = ', '.join(lab_items.keys())
query = f"""
SELECT le.subject_id, le.hadm_id, i.stay_id,
le.itemid, le.charttime, le.valuenum
FROM mimiciv_hosp.labevents le
INNER JOIN mimiciv_icu.icustays i ON le.subject_id = i.subject_id
AND le.charttime BETWEEN i.intime - INTERVAL '24 HOURS' AND i.outtime + INTERVAL '24 HOURS'
WHERE i.stay_id IN {stay_ids_str}
AND le.itemid IN ({item_ids_str})
AND le.valuenum IS NOT NULL
ORDER BY le.subject_id, le.hadm_id, le.charttime
"""
lab_df = pd.read_sql(query, engine)
# 添加检查项目名称
lab_df['lab_name'] = lab_df['itemid'].astype(str).map(lab_items)
print(f"提取到 {len(lab_df)} 条实验室检查记录")
return lab_df
# 提取SOFA评分相关数据
def extract_sofa_data(engine, sepsis_df):
"""
提取脓毒症患者的SOFA评分数据
参数:
engine: SQLAlchemy数据库引擎
sepsis_df: 脓毒症患者DataFrame
返回:
sofa_df: SOFA评分数据
"""
# 获取所有脓毒症患者的stay_id列表
stay_ids = tuple(sepsis_df['stay_id'].unique())
# 如果只有一个ID,需要特殊处理tuple语法
if len(stay_ids) == 1:
stay_ids_str = f"({stay_ids[0]})"
else:
stay_ids_str = str(stay_ids)
query = f"""
SELECT *
FROM mimiciv_derived.sofa
WHERE stay_id IN {stay_ids_str}
ORDER BY stay_id, starttime
"""
sofa_df = pd.read_sql(query, engine)
print(f"提取到 {len(sofa_df)} 条SOFA评分记录")
return sofa_df
# 提取药物使用数据
# 修改后的药物使用数据提取函数
def extract_medication_data(engine, sepsis_df):
"""
提取脓毒症患者的药物使用数据,重点是抗生素、升压药等
"""
# 首先查看prescriptions表的实际列结构
query = """
SELECT column_name
FROM information_schema.columns
WHERE table_schema = 'mimiciv_hosp'
AND table_name = 'prescriptions'
"""
columns_df = pd.read_sql(query, engine)
print("prescriptions表的列名:", columns_df['column_name'].tolist())
# 获取stay_id列表
stay_ids = tuple(sepsis_df['stay_id'].unique())
# 如果只有一个ID,需要特殊处理tuple语法
if len(stay_ids) == 1:
stay_ids_str = f"({stay_ids[0]})"
else:
stay_ids_str = str(stay_ids)
# 使用简化版查询,只包含基本列
query = f"""
SELECT p.subject_id, p.hadm_id, p.starttime, p.stoptime,
p.drug, p.route, p.dose_val_rx, p.dose_unit_rx
FROM mimiciv_hosp.prescriptions p
JOIN mimiciv_icu.icustays i ON p.hadm_id = i.hadm_id
WHERE i.stay_id IN {stay_ids_str}
ORDER BY p.subject_id, p.hadm_id, p.starttime
"""
med_df = pd.read_sql(query, engine)
print(f"提取到 {len(med_df)} 条药物使用记录")
return med_df
# 提取诊断信息
# 修改后的诊断信息提取函数
def extract_diagnosis_data(engine, sepsis_df):
"""
提取脓毒症患者的诊断信息
"""
# 首先确认icustays和hadm_id的关系
if 'hadm_id' not in sepsis_df.columns:
# 获取hadm_id
query = f"""
SELECT DISTINCT i.stay_id, i.hadm_id
FROM mimiciv_icu.icustays i
WHERE i.stay_id IN {tuple(sepsis_df['stay_id'].unique())}
"""
stay_hadm_map = pd.read_sql(query, engine)
sepsis_with_hadm = sepsis_df.merge(stay_hadm_map, on='stay_id', how='left')
hadm_ids = tuple(stay_hadm_map['hadm_id'].unique())
else:
hadm_ids = tuple(sepsis_df['hadm_id'].unique())
# 如果只有一个ID,需要特殊处理tuple语法
if len(hadm_ids) == 1:
hadm_ids_str = f"({hadm_ids[0]})"
else:
hadm_ids_str = str(hadm_ids)
# 简化的诊断查询
query = f"""
SELECT d.subject_id, d.hadm_id, d.seq_num,
d.icd_code, d.icd_version
FROM mimiciv_hosp.diagnoses_icd d
WHERE d.hadm_id IN {hadm_ids_str}
ORDER BY d.subject_id, d.hadm_id, d.seq_num
"""
diag_df = pd.read_sql(query, engine)
# 尝试添加诊断名称(如果存在d_icd_diagnoses表)
try:
diagnoses_name_query = """
SELECT DISTINCT icd_code, icd_version, long_title
FROM mimiciv_hosp.d_icd_diagnoses
"""
icd_names = pd.read_sql(diagnoses_name_query, engine)
diag_df = diag_df.merge(icd_names, on=['icd_code', 'icd_version'], how='left')
except:
print("无法获取诊断名称,将只包含ICD代码")
print(f"提取到 {len(diag_df)} 条诊断记录")
return diag_df
# 合并数据并初步处理
def process_data(sepsis_df, plt_df, lab_df, sofa_df, med_df, diag_df):
"""合并所有提取的数据并进行初步处理"""
# 1. 标记血小板减少患者 (血小板计数 < 150 x 10^9/L)
patients_with_plt = plt_df['stay_id'].unique()
sepsis_with_plt = sepsis_df[sepsis_df['stay_id'].isin(patients_with_plt)].copy()
# 检查sepsis_df是否包含hadm_id列
if 'hadm_id' not in sepsis_df.columns:
print("警告: sepsis_df中不存在hadm_id列,将从icustays表中获取")
# 从plt_df中提取stay_id和hadm_id的映射
stay_hadm_map = plt_df[['stay_id', 'hadm_id']].drop_duplicates()
# 将hadm_id添加到sepsis_with_plt
sepsis_with_plt = sepsis_with_plt.merge(stay_hadm_map, on='stay_id', how='left')
# 对每个患者计算血小板相关特征
plt_features = []
for stay_id in patients_with_plt:
patient_plt = plt_df[plt_df['stay_id'] == stay_id].sort_values('charttime')
if len(patient_plt) == 0:
continue
# 基线血小板计数(首次测量值)
baseline_plt = patient_plt.iloc[0]['platelet_count']
# 最低血小板计数
min_plt = patient_plt['platelet_count'].min()
min_plt_time = patient_plt.loc[patient_plt['platelet_count'].idxmin(), 'charttime']
# 最后一次血小板计数
last_plt = patient_plt.iloc[-1]['platelet_count']
# 血小板减少判定 (< 150 x 10^9/L)
is_thrombocytopenia = 1 if min_plt < 150 else 0
# 严重血小板减少判定 (< 50 x 10^9/L)
is_severe_thrombocytopenia = 1 if min_plt < 50 else 0
# 血小板下降幅度(相对于基线)
plt_decrease_pct = (baseline_plt - min_plt) / baseline_plt * 100 if baseline_plt > 0 else 0
# 血小板是否恢复(最后一次测量值比最低值高50%以上)
plt_recovery = 1 if last_plt > min_plt * 1.5 else 0
# 测量次数
plt_measurements = len(patient_plt)
# 组装特征
plt_features.append({
'stay_id': stay_id,
'subject_id': patient_plt.iloc[0]['subject_id'],
'hadm_id': patient_plt.iloc[0]['hadm_id'],
'baseline_plt': baseline_plt,
'min_plt': min_plt,
'min_plt_time': min_plt_time,
'last_plt': last_plt,
'is_thrombocytopenia': is_thrombocytopenia,
'is_severe_thrombocytopenia': is_severe_thrombocytopenia,
'plt_decrease_pct': plt_decrease_pct,
'plt_recovery': plt_recovery,
'plt_measurements': plt_measurements
})
# 创建血小板特征DataFrame
plt_features_df = pd.DataFrame(plt_features)
# 根据available的列进行合并
merge_cols = ['stay_id', 'subject_id']
if 'hadm_id' in sepsis_with_plt.columns:
merge_cols.append('hadm_id')
# 合并血小板特征与患者基本信息
sepsis_plt_df = sepsis_with_plt.merge(plt_features_df, on=merge_cols, how='inner')
# 计算患者死亡率
mortality_rate = sepsis_plt_df['hospital_expire_flag'].mean() * 100
# 血小板减少的患者比例
thrombocytopenia_rate = sepsis_plt_df['is_thrombocytopenia'].mean() * 100
# 严重血小板减少的患者比例
severe_thrombocytopenia_rate = sepsis_plt_df['is_severe_thrombocytopenia'].mean() * 100
print(f"合并后数据集包含 {len(sepsis_plt_df)} 名患者")
print(f"医院死亡率: {mortality_rate:.2f}%")
print(f"血小板减少(<150×10^9/L)比例: {thrombocytopenia_rate:.2f}%")
print(f"严重血小板减少(<50×10^9/L)比例: {severe_thrombocytopenia_rate:.2f}%")
# 返回处理后的数据
processed_data = {
'sepsis_plt_df': sepsis_plt_df,
'plt_df': plt_df,
'lab_df': lab_df,
'sofa_df': sofa_df,
'med_df': med_df,
'diag_df': diag_df
}
return processed_data
# 保存提取的数据
def save_data(processed_data, output_dir='data'):
"""
将处理后的数据保存到指定目录
参数:
processed_data: 处理后的数据字典
output_dir: 输出目录
"""
# 创建输出目录(如果不存在)
if not os.path.exists(output_dir):
os.makedirs(output_dir)
# 保存各数据集
for name, df in processed_data.items():
output_path = os.path.join(output_dir, f"{name}.csv")
df.to_csv(output_path, index=False)
print(f"已保存 {name} 到 {output_path}")
# 主函数
def main():
# 连接数据库
engine, conn = connect_to_postgres()
try:
# 提取脓毒症患者队列
sepsis_df = extract_sepsis_cohort(engine)
# 提取血小板计数数据
plt_df = extract_platelet_data(engine, sepsis_df)
# 提取其他实验室检查数据
lab_df = extract_lab_data(engine, sepsis_df)
# 提取SOFA评分数据
sofa_df = extract_sofa_data(engine, sepsis_df)
# 提取药物使用数据
med_df = extract_medication_data(engine, sepsis_df)
# 提取诊断信息
diag_df = extract_diagnosis_data(engine, sepsis_df)
# 处理数据
processed_data = process_data(sepsis_df, plt_df, lab_df, sofa_df, med_df, diag_df)
# 保存数据
save_data(processed_data)
print("数据提取和处理完成!")
finally:
# 关闭数据库连接
conn.close()
# 运行主函数
if __name__ == "__main__":
main()
else:
# 在Jupyter Notebook中执行时
# 导入必要的库
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sqlalchemy import create_engine
import psycopg2
import os
from datetime import datetime, timedelta
import warnings
warnings.filterwarnings('ignore')
# 设置绘图风格
plt.style.use('ggplot')
sns.set(style="whitegrid")
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文
plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号
数据提取解释:
数据提取与纳入排除步骤
- 脓毒症患者队列提取 数据来源: 使用MIMIC-IV数据库中的mimiciv_derived.sepsis3表,该表基于Sepsis-3国际共识定义 关联表格: patients表获取人口学特征(性别、年龄) admissions表获取住院信息(入院/出院时间、死亡标志) icustays表获取ICU住院信息(入/出ICU时间、住院时长) 纳入标准: 所有在sepsis3表中的患者 排除标准: 无特定排除标准 队列规模: 成功提取41,295名脓毒症患者
- 血小板计数数据提取 数据来源: labevents表中的血小板计数(ITEMID=51265) 时间窗口: ICU入院前24小时至出院后24小时 排除标准: 空值(NULL) 异常值(≤0或≥1000×10^9/L) 数据规模: 提取441,130条血小板计数记录,涉及41,273位患者
- 其他实验室检查数据提取 数据来源: labevents表中的19种关键实验室检查 检查项目: 肌酐、胆红素、电解质、白细胞计数、凝血功能等 时间窗口: 与血小板计数相同 排除标准: 空值 数据规模: 提取5,756,096条实验室检查记录
- SOFA评分数据提取 数据来源: mimiciv_derived.sofa表 纳入标准: 所有脓毒症患者的SOFA评分记录 数据规模: 提取5,518,962条SOFA评分记录
- 药物使用数据提取 数据来源: prescriptions表 药物信息: 药物名称、给药途径、剂量等 数据规模: 提取6,108,555条药物使用记录
- 诊断信息提取 数据来源: diagnoses_icd表和d_icd_diagnoses表 提取内容: ICD编码及其对应的诊断名称 数据规模: 提取822,716条诊断记录
- 数据合并与特征计算 主要步骤: 将脓毒症患者与血小板数据合并 计算血小板相关特征: 基线血小板计数(首次测量值) 最低血小板计数及其时间 血小板减少判定(<150×10^9/L) 严重血小板减少判定(<50×10^9/L) 血小板下降幅度(相对基线的百分比) 血小板恢复情况(最后测量值比最低值高50%以上) 最终分析集: 21,838名有完整血小板数据的脓毒症患者
保存的CSV文件内容
- sepsis_plt_df.csv 核心分析数据集,包含脓毒症患者基本信息与血小板特征 主要字段: 患者ID(subject_id, stay_id, hadm_id) 人口学特征(性别、年龄) 住院信息(入院/出院时间、死亡标志) ICU信息(入/出ICU时间、住院天数) 血小板特征(基线值、最低值、血小板减少状态等) 关键统计: 医院死亡率: 18.61% 血小板减少(<150×10^9/L)比例: 58.52% 严重血小板减少(<50×10^9/L)比例: 11.54%
- plt_df.csv 原始血小板计数数据 主要字段: 患者ID、测量时间、血小板计数值 记录数: 441,130条
- lab_df.csv 其他实验室检查数据 主要字段: 患者ID、检测项目、检测时间、检测值 记录数: 5,756,096条
- sofa_df.csv SOFA评分数据 主要字段: 患者ID、评分时间、总分及各系统分项评分 记录数: 5,518,962条
- med_df.csv 药物使用数据 主要字段: 患者ID、用药时间、药物名称、给药途径、剂量 记录数: 6,108,555条
- diag_df.csv 诊断信息数据 主要字段: 患者ID、诊断序号、ICD编码及版本、诊断名称 记录数: 822,716条
数据初步分析
描述性统计分析
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
# 加载保存的数据
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import os
from scipy import stats
# 设置中文显示
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
# 加载保存的数据
data_dir = 'data'
sepsis_plt_df = pd.read_csv(os.path.join(data_dir, 'sepsis_plt_df.csv'))
# 基本统计
print(f"总患者数: {len(sepsis_plt_df)}")
print(f"血小板减少患者数: {sepsis_plt_df['is_thrombocytopenia'].sum()} ({sepsis_plt_df['is_thrombocytopenia'].mean()*100:.2f}%)")
print(f"严重血小板减少患者数: {sepsis_plt_df['is_severe_thrombocytopenia'].sum()} ({sepsis_plt_df['is_severe_thrombocytopenia'].mean()*100:.2f}%)")
# 人口学特征
print("\n==== 人口学特征 ====")
print(f"平均年龄: {sepsis_plt_df['age'].mean():.2f} ± {sepsis_plt_df['age'].std():.2f}")
print(f"性别分布: 男性 {(sepsis_plt_df['gender']=='M').mean()*100:.2f}%, 女性 {(sepsis_plt_df['gender']=='F').mean()*100:.2f}%")
# 住院情况
sepsis_plt_df['los_days'] = sepsis_plt_df['icu_los'] # ICU住院天数
print(f"\n平均ICU住院天数: {sepsis_plt_df['los_days'].mean():.2f} ± {sepsis_plt_df['los_days'].std():.2f}")
print(f"医院死亡率: {sepsis_plt_df['hospital_expire_flag'].mean()*100:.2f}%")
总患者数: 21838
血小板减少患者数: 12780 (58.52%)
严重血小板减少患者数: 2520 (11.54%)
==== 人口学特征 ====
平均年龄: 64.58 ± 14.98
性别分布: 男性 59.26%, 女性 40.74%
平均ICU住院天数: 5.98 ± 8.00 医院死亡率: 18.61%
分组比较分析.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
# 创建分组
thromb_group = sepsis_plt_df[sepsis_plt_df['is_thrombocytopenia']==1]
non_thromb_group = sepsis_plt_df[sepsis_plt_df['is_thrombocytopenia']==0]
# 创建比较表
comparison = pd.DataFrame({
'特征': ['患者数', '年龄', '男性比例(%)', 'ICU住院天数', '死亡率(%)'],
'血小板减少组': [
len(thromb_group),
f"{thromb_group['age'].mean():.2f} ± {thromb_group['age'].std():.2f}",
f"{(thromb_group['gender']=='M').mean()*100:.2f}",
f"{thromb_group['los_days'].mean():.2f} ± {thromb_group['los_days'].std():.2f}",
f"{thromb_group['hospital_expire_flag'].mean()*100:.2f}"
],
'非血小板减少组': [
len(non_thromb_group),
f"{non_thromb_group['age'].mean():.2f} ± {non_thromb_group['age'].std():.2f}",
f"{(non_thromb_group['gender']=='M').mean()*100:.2f}",
f"{non_thromb_group['los_days'].mean():.2f} ± {non_thromb_group['los_days'].std():.2f}",
f"{non_thromb_group['hospital_expire_flag'].mean()*100:.2f}"
],
'p值': [
'-',
f"{stats.ttest_ind(thromb_group['age'].dropna(), non_thromb_group['age'].dropna()).pvalue:.4f}",
f"{stats.chi2_contingency([[sum(thromb_group['gender']=='M'), sum(thromb_group['gender']=='F')], [sum(non_thromb_group['gender']=='M'), sum(non_thromb_group['gender']=='F')]])[1]:.4f}",
f"{stats.ttest_ind(thromb_group['los_days'].dropna(), non_thromb_group['los_days'].dropna()).pvalue:.4f}",
f"{stats.chi2_contingency([[sum(thromb_group['hospital_expire_flag']==1), sum(thromb_group['hospital_expire_flag']==0)], [sum(non_thromb_group['hospital_expire_flag']==1), sum(non_thromb_group['hospital_expire_flag']==0)]])[1]:.4f}"
]
})
print(comparison)
| 特征 | 血小板减少组 | 非血小板减少组 | p值 |
|---|---|---|---|
| 患者数 | 12780 | 9058 | - |
| 年龄 | 64.34 ± 14.66 | 64.92 ± 15.42 | 0.0047 |
| 男性比例(%) | 62.43 | 54.79 | 0.0000 |
| ICU住院天数 | 6.47 ± 8.80 | 5.30 ± 6.65 | 0.0000 |
| 死亡率(%) | 21.05 | 15.16 | 0.0000 |
血小板减少特征详细分析
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
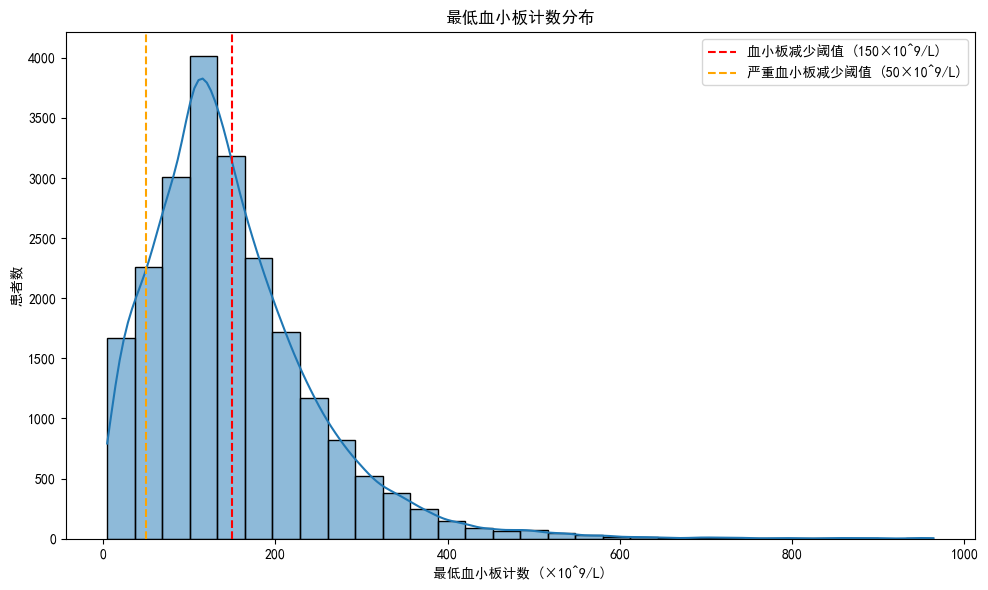
# 血小板减少的严重程度分布
plt.figure(figsize=(10, 6))
sns.histplot(data=sepsis_plt_df, x='min_plt', bins=30, kde=True)
plt.axvline(x=150, color='r', linestyle='--', label='血小板减少阈值 (150×10^9/L)')
plt.axvline(x=50, color='orange', linestyle='--', label='严重血小板减少阈值 (50×10^9/L)')
plt.title('最低血小板计数分布')
plt.xlabel('最低血小板计数 (×10^9/L)')
plt.ylabel('患者数')
plt.legend()
plt.tight_layout()
plt.show()
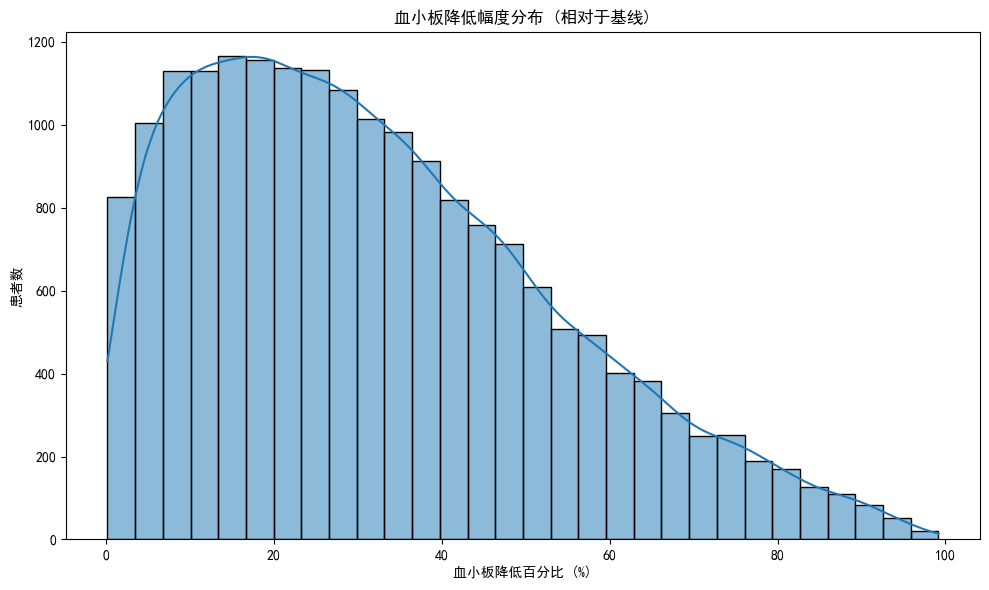
# 血小板降低幅度分析
plt.figure(figsize=(10, 6))
sns.histplot(data=sepsis_plt_df[sepsis_plt_df['plt_decrease_pct']>0],
x='plt_decrease_pct', bins=30, kde=True)
plt.title('血小板降低幅度分布 (相对于基线)')
plt.xlabel('血小板降低百分比 (%)')
plt.ylabel('患者数')
plt.tight_layout()
plt.show()
# 血小板恢复率
recovery_rate = sepsis_plt_df[sepsis_plt_df['is_thrombocytopenia']==1]['plt_recovery'].mean() * 100
print(f"血小板减少患者中的恢复率: {recovery_rate:.2f}%")
血小板减少患者中的恢复率: 39.84%
血小板减少与临床结局关系
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
# 创建严重程度分组
sepsis_plt_df['plt_group'] = pd.cut(
sepsis_plt_df['min_plt'],
bins=[0, 50, 100, 150, 1000],
labels=['<50', '50-100', '100-150', '>150']
)
# 各组死亡率
mortality_by_group = sepsis_plt_df.groupby('plt_group')['hospital_expire_flag'].mean() * 100
print("各血小板组的死亡率:")
print(mortality_by_group)
# 可视化死亡率
plt.figure(figsize=(10, 6))
sns.barplot(x=mortality_by_group.index, y=mortality_by_group.values)
plt.title('不同血小板水平组的死亡率')
plt.xlabel('最低血小板计数 (×10^9/L)')
plt.ylabel('死亡率 (%)')
plt.tight_layout()
plt.show()
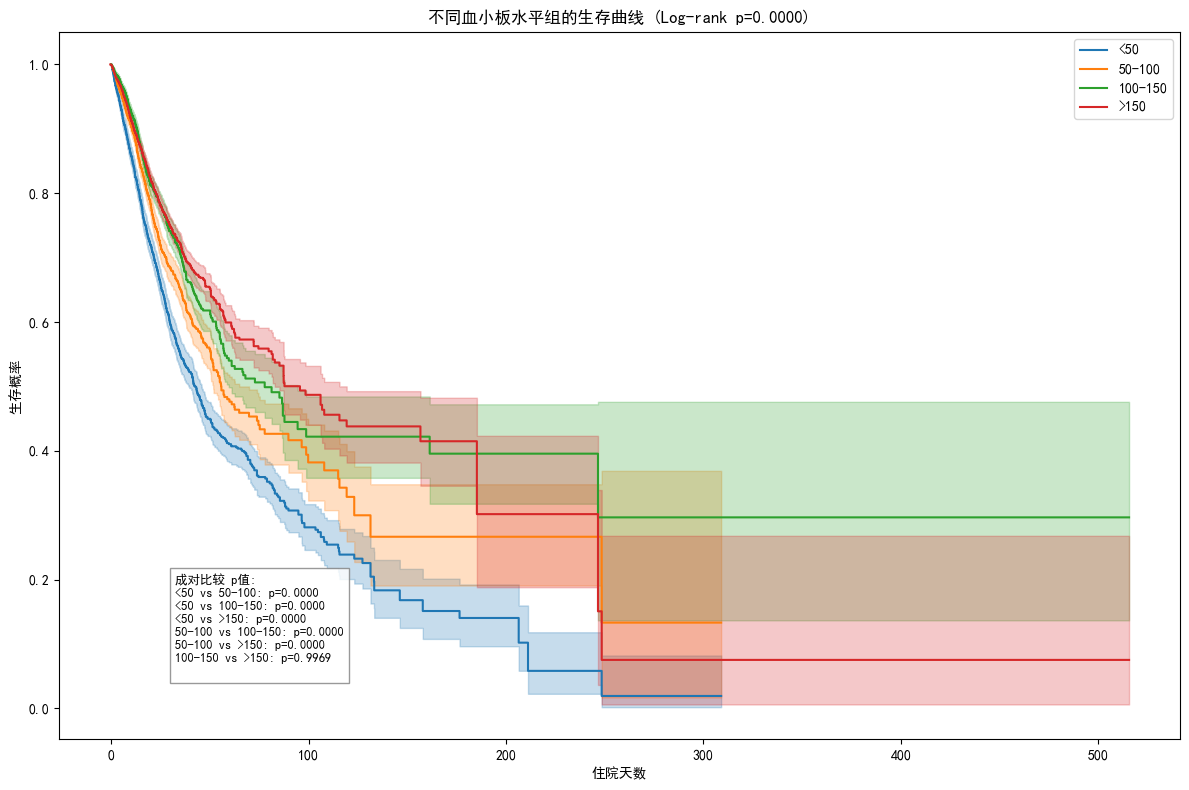
# 生存曲线
try:
from lifelines import KaplanMeierFitter
from lifelines.statistics import logrank_test, multivariate_logrank_test
kmf = KaplanMeierFitter()
# 计算住院时长
sepsis_plt_df['hosp_los'] = (pd.to_datetime(sepsis_plt_df['dischtime']) -
pd.to_datetime(sepsis_plt_df['admittime'])).dt.total_seconds() / (24*3600)
plt.figure(figsize=(12, 8))
# 存储各组数据用于统计检验
groups = {}
for name, group in sepsis_plt_df.groupby('plt_group'):
kmf.fit(group['hosp_los'], event_observed=group['hospital_expire_flag'], label=name)
kmf.plot()
groups[name] = (group['hosp_los'], group['hospital_expire_flag'])
# 执行多组log-rank检验 (修复部分)
# 将所有组的数据堆叠到一起
durations = []
events = []
group_labels = []
for name, (duration, event) in groups.items():
durations.extend(duration.values) # 转换为数组或列表
events.extend(event.values) # 转换为数组或列表
group_labels.extend([name] * len(duration))
# 使用堆叠的数据进行log-rank测试
results = multivariate_logrank_test(
np.array(durations),
np.array(group_labels),
np.array(events)
)
p_value = results.p_value
# 添加p值到标题
plt.title(f'不同血小板水平组的生存曲线 (Log-rank p={p_value:.4f})')
plt.xlabel('住院天数')
plt.ylabel('生存概率')
# 添加成对比较p值
group_names = list(groups.keys())
p_values_text = "成对比较 p值:\n"
for i in range(len(group_names)):
for j in range(i+1, len(group_names)):
g1_name = group_names[i]
g2_name = group_names[j]
T1, E1 = groups[g1_name]
T2, E2 = groups[g2_name]
# 成对log-rank检验
lr_result = logrank_test(T1, T2, E1, E2)
p_values_text += f"{g1_name} vs {g2_name}: p={lr_result.p_value:.4f}\n"
# 将p值文本添加到图表中
plt.figtext(0.15, 0.15, p_values_text, fontsize=9,
bbox=dict(facecolor='white', alpha=0.8, edgecolor='gray'))
plt.tight_layout()
plt.show()
except ImportError:
print("需要安装lifelines包进行生存分析: pip install lifelines")
各血小板组的死亡率:
| plt_group | % |
|---|---|
| <50 | 42.441860 |
| 50-100 | 19.347426 |
| 100-150 | 12.943949 |
| >150 | 15.136642 |
研究药物与血小板减少的关系
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
# 加载药物数据
med_df = pd.read_csv(os.path.join(data_dir, 'med_df.csv'))
# 首先检查med_df的列
print("药物数据集的列名:", med_df.columns.tolist())
# 创建从hadm_id到stay_id的映射
hadm_to_stay = sepsis_plt_df[['hadm_id', 'stay_id']].drop_duplicates()
# 提取常见抗生素和其他关键药物
common_drugs = [
'vancomycin', 'ceftriaxone', 'piperacillin', 'tazobactam',
'meropenem', 'ciprofloxacin', 'metronidazole', 'norepinephrine',
'epinephrine', 'vasopressin', 'heparin', 'enoxaparin'
]
# 分析每种药物与血小板减少的关系
drug_analysis = []
for drug in common_drugs:
# 找出使用该药物的患者
# 首先通过hadm_id找到使用该药物的患者
drug_hadm_ids = med_df[med_df['drug'].str.contains(drug, case=False, na=False)]['hadm_id'].unique()
# 然后通过hadm_id找到对应的stay_id
matched_stays = hadm_to_stay[hadm_to_stay['hadm_id'].isin(drug_hadm_ids)]
drug_users = matched_stays['stay_id'].unique()
# 计算使用该药物的患者中血小板减少的比例
drug_users_data = sepsis_plt_df[sepsis_plt_df['stay_id'].isin(drug_users)]
non_users_data = sepsis_plt_df[~sepsis_plt_df['stay_id'].isin(drug_users)]
if len(drug_users_data) > 0 and len(non_users_data) > 0:
thromb_rate_users = drug_users_data['is_thrombocytopenia'].mean() * 100
thromb_rate_non_users = non_users_data['is_thrombocytopenia'].mean() * 100
# 计算统计显著性
contingency = [
[sum(drug_users_data['is_thrombocytopenia']==1), sum(drug_users_data['is_thrombocytopenia']==0)],
[sum(non_users_data['is_thrombocytopenia']==1), sum(non_users_data['is_thrombocytopenia']==0)]
]
_, p_value, _, _ = stats.chi2_contingency(contingency)
drug_analysis.append({
'药物': drug,
'使用人数': len(drug_users_data),
'血小板减少率(%)': thromb_rate_users,
'未使用人数': len(non_users_data),
'未使用血小板减少率(%)': thromb_rate_non_users,
'差异(%)': thromb_rate_users - thromb_rate_non_users,
'p值': p_value
})
# 显示结果
drug_analysis_df = pd.DataFrame(drug_analysis)
print(drug_analysis_df.sort_values('差异(%)', ascending=False))
# 可视化结果
plt.figure(figsize=(12, 8))
sorted_df = drug_analysis_df.sort_values('差异(%)', ascending=False)
sns.barplot(x='药物', y='差异(%)', data=sorted_df)
plt.title('各种药物使用与血小板减少的关联')
plt.xlabel('药物')
plt.ylabel('血小板减少率差异 (%)')
plt.xticks(rotation=45)
plt.tight_layout()
plt.show()
| 药物 | 使用人数 | 血小板减少率(%) | 未使用人数 | 未使用血小板减少率(%) | 差异(%) | p值 |
|---|---|---|---|---|---|---|
| vasopressin | 4345 | 73.578826 | 17493 | 54.781913 | 18.796913 | 5.098236e-112 |
| epinephrine | 10029 | 65.549905 | 11809 | 52.553137 | 12.996768 | 6.019883e-84 |
| norepinephrine | 8914 | 65.111061 | 12924 | 53.977097 | 11.133964 | 1.917429e-60 |
| meropenem | 3410 | 63.167155 | 18428 | 57.662253 | 5.504902 | 2.304819e-09 |
| ciprofloxacin | 4909 | 58.545529 | 16929 | 58.514974 | 0.030554 | 9.826029e-01 |
| vancomycin | 16126 | 58.216545 | 5712 | 59.383754 | -1.167209 | 1.277607e-01 |
| ceftriaxone | 6084 | 56.689678 | 15754 | 59.229402 | -2.539724 | 6.744361e-04 |
| metronidazole | 6807 | 56.662259 | 15031 | 59.363981 | -2.701722 | 1.849884e-04 |
| piperacillin | 6309 | 56.522428 | 15529 | 59.334149 | -2.811721 | 1.404570e-04 |
| tazobactam | 6330 | 56.477093 | 15508 | 59.356461 | -2.879368 | 9.492910e-05 |
| enoxaparin | 2424 | 50.123762 | 19414 | 59.570413 | -9.446651 | 6.753285e-19 |
| heparin | 19310 | 57.115484 | 2528 | 69.264241 | -12.148756 | 2.676747e-31 |
对血小板减少亚型聚类分析
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
# 提取血小板动态特征
patients = sepsis_plt_df['stay_id'].unique()
plt_patterns = []
for stay_id in patients:
patient_plt = plt_df[plt_df['stay_id'] == stay_id].sort_values('charttime')
if len(patient_plt) >= 3: # 至少有3次测量
# 转换为相对时间
patient_plt['charttime'] = pd.to_datetime(patient_plt['charttime'])
patient_plt['days'] = (patient_plt['charttime'] - patient_plt['charttime'].min()).dt.total_seconds() / (24*3600)
# 提取特征
first_plt = patient_plt.iloc[0]['platelet_count']
min_plt = patient_plt['platelet_count'].min()
last_plt = patient_plt.iloc[-1]['platelet_count']
time_to_min = patient_plt.loc[patient_plt['platelet_count'].idxmin(), 'days']
# 计算变化率
if len(patient_plt) > 1:
slopes = []
for i in range(1, len(patient_plt)):
dt = patient_plt.iloc[i]['days'] - patient_plt.iloc[i-1]['days']
if dt > 0:
dp = patient_plt.iloc[i]['platelet_count'] - patient_plt.iloc[i-1]['platelet_count']
slopes.append(dp/dt)
mean_slope = np.mean(slopes) if slopes else 0
else:
mean_slope = 0
plt_patterns.append({
'stay_id': stay_id,
'first_plt': first_plt,
'min_plt': min_plt,
'last_plt': last_plt,
'time_to_min': time_to_min,
'mean_slope': mean_slope,
'recovery_ratio': last_plt/min_plt if min_plt > 0 else 1,
'measurements': len(patient_plt)
})
plt_patterns_df = pd.DataFrame(plt_patterns)
# 使用K-means聚类识别血小板减少模式
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import KMeans
# 选择聚类特征
cluster_features = ['first_plt', 'min_plt', 'last_plt', 'time_to_min', 'mean_slope', 'recovery_ratio']
X_cluster = plt_patterns_df[cluster_features].copy()
# 数据标准化
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X_cluster.fillna(X_cluster.mean()))
# 确定最佳聚类数
from sklearn.metrics import silhouette_score
silhouette_scores = []
for k in range(2, 8):
kmeans = KMeans(n_clusters=k, random_state=42)
clusters = kmeans.fit_predict(X_scaled)
silhouette_scores.append(silhouette_score(X_scaled, clusters))
# 绘制轮廓系数
plt.figure(figsize=(10, 6))
plt.plot(range(2, 8), silhouette_scores, marker='o')
plt.title('轮廓系数与聚类数的关系')
plt.xlabel('聚类数')
plt.ylabel('轮廓系数')
plt.grid(True)
plt.show()
# 基于最佳聚类数进行聚类
best_k = np.argmax(silhouette_scores) + 2
kmeans = KMeans(n_clusters=best_k, random_state=42)
plt_patterns_df['cluster'] = kmeans.fit_predict(X_scaled)
# 分析各聚类的特征
cluster_profile = plt_patterns_df.groupby('cluster')[cluster_features].mean()
print("各聚类的特征概况:")
print(cluster_profile)
# 可视化各聚类的血小板变化模式
plt.figure(figsize=(14, 10))
for cluster in range(best_k):
cluster_patients = plt_patterns_df[plt_patterns_df['cluster']==cluster]['stay_id'].sample(min(5, sum(plt_patterns_df['cluster']==cluster))).tolist()
plt.subplot(best_k, 1, cluster+1)
for i, stay_id in enumerate(cluster_patients):
patient_plt = plt_df[plt_df['stay_id'] == stay_id].copy()
patient_plt['charttime'] = pd.to_datetime(patient_plt['charttime'])
patient_plt['days'] = (patient_plt['charttime'] - patient_plt['charttime'].min()).dt.total_seconds() / (24*3600)
plt.plot(patient_plt['days'], patient_plt['platelet_count'], marker='o', label=f'患者 {i+1}')
plt.axhline(y=150, color='r', linestyle='--', label='血小板减少阈值')
plt.axhline(y=50, color='orange', linestyle='--', label='严重血小板减少阈值')
plt.title(f'聚类 {cluster+1} 的血小板变化模式')
plt.xlabel('天数')
plt.ylabel('血小板计数 (×10^9/L)')
plt.legend()
plt.grid(True)
plt.tight_layout()
plt.show()
# 分析各聚类的临床结局
# 将聚类信息合并回主数据集
sepsis_cluster = sepsis_plt_df.merge(plt_patterns_df[['stay_id', 'cluster']], on='stay_id', how='inner')
mortality_by_cluster = sepsis_cluster.groupby('cluster')['hospital_expire_flag'].mean() * 100
print("各聚类的死亡率:")
print(mortality_by_cluster)
各聚类的特征概况:
| cluster | first_plt | min_plt | last_plt | time_to_min | mean_slope | recovery_ratio |
|---|---|---|---|---|---|---|
| 0 | 218.102210 | 83.399171 | 214.726519 | 17.195416 | 39.935725 | 5.837524 |
| 1 | 348.021392 | 261.797475 | 370.828862 | 2.250703 | -5.449554 | 1.573270 |
| 2 | 158.889035 | 107.530784 | 153.654326 | 2.092474 | 4.901084 | 1.610446 |
各聚类的死亡率:
| cluster | % |
|---|---|
| 0 | 37.845304 |
| 1 | 14.395932 |
| 2 | 18.639153 |
关键点解析
不同血小板水平组的死亡率存在明显差异,尤其是<50组的死亡率显著高于其他组
药物分析发现部分药物使用与血小板减少存在相关性,但是药物使用与疾病严重程度必然相关,又回到了 疾病严重与血小板下降 孰因孰果的问题,无法得出因果关系
血小板减少亚型聚类分析发现了三个亚群,聚类0的死亡率(37.8%)是聚类1(14.4%)的2.6倍,聚类0达到最低点时间明显更长
总结与思考
这个问题的核心是…,通过…方法可以有效解决。